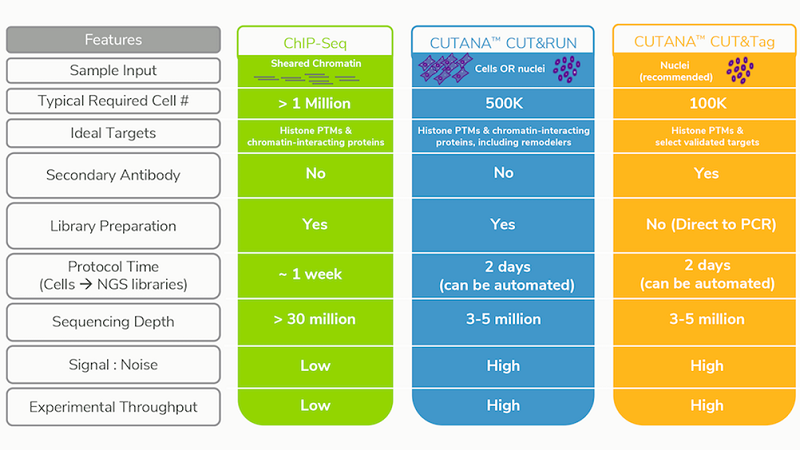
1. CUT&RUN 분석은 어떤 장점이 있나요?
CUT&RUN assay는 전사인자와 크로마틴 관련 단백질의 상호작용을 분석하는 데 사용되는 기법입니다. 기존에 가장 많이 사용되는 기법으로는 ChIP-Seq이 있으나, 시간 집약적이고 많은 양의 세포를 요구하며, 백그라운드가 높아 signal-to-noise ratio가 낮고, 불용성 크로마틴 단백질은 호환되지 않는 등의 단점이 있었습니다. CUT&RUN은 ChIC(Chromatin ImmunoCleavage) 기법을 활용하여, 시간을 단축하고 더 적은 양의 세포로 더 높은 해상도와 민감도를 보이는 결과를 보여주는 최신 기술입니다. 아래 표를 참고하면, CUT&RUN의 장점을 더 많이 알 수 있습니다.
2. CUT&RUN 기술의 작동방식을 설명해주세요
세포(또는 핵)를 고체 지지대에 고정하고 투과화되게 한 뒤, 히스톤 PTM 또는 크로마틴과 상호작용하는 단백질에 항체를 추가합니다. 타겟-DNA 복합체를 절단하기 위해 pAG-MNase를 추가하고 활성화한 뒤, 타겟-DNA 복합체가 확산하면 상층액을 수집하여 DNA를 추출하여 시퀀싱할 라이브러리를 준비합니다. 준비된 라이브러리를 활용하여, 차세대 시퀀싱(NGS)과 데이터 분석을 합니다.
3. ChIP을 하기에 세포가 부족할 때 어떻게 해야 하나요?
ChIP은 일반적으로, 100만개 이상의 세포가 있을 때 분석이 가능하지만, CUTANA CUT&RUN과 CUT&Tag은 각각 50만개, 10만개의 세포만을 요구합니다. 또한 Hainer et al.에 따르면, CUTANA CUT&RUN은 적게는 5,000개의 세포로도 분석이 가능합니다.
4. CUT&Tag을 사용하기 위해서는 2차 항체가 필요한데, CUT&RUN도 2차 항체가 필요한가요?
(일반적으로는) 필요하지 않습니다. CUT&Tag은 라이브러리를 생성하지 않고, Tn5를 이용하여 직접적으로 PCR을 이용하는 기법입니다. Tn5를 부착하기 위해 2차 항체가 필요합니다. 하지만, CUT&RUN은 Tn5를 필요로 하지 않고 별도로 라이브러리를 제작하기 때문에 Tn5를 부착하기 위한 2차 항체를 요구하지 않습니다.
5. CUT&RUN을 DNA binding protein이 아닌 RNA binding protein에도 사용할 수 있나요?
네, 가능합니다. 프로토콜의 일부를 변형하여 사용할 수 있습니다. 사용한 논문은 다음과 같습니다.
Daneshvar K, et al. lncRNA DIGIT and BRD3 protein form phase-separated condensates to regulate endoderm differentiation. bioRxiv, 2019.
6. CUT&Tag이 더 빠른데, CUT&RUN을 사용해야 하는 이유가 있나요?
CUT&Tag이 라이브러리를 생성하지 않고, 직접적으로 PCR을 하기 때문에 속도 측면에서의 장점이 있지만, CUT&RUN은 그 외에 다양한 장점이 있습니다.
우선, CUT&RUN은 정확도가 높습니다. CUT&Tag의 장점인 Tn5를 사용하여 라이브러리 생성 없이 바로 PCR을 하는 것은 off-target Tn5 activity를 야기할 수도 있습니다. 따라서, signal-to-ratio는 CUT&RUN이 높아 CUT&RUN을 이용하시면, 더욱 민감하고 해상도가 높은 분석 결과를 얻을 수 있습니다.
또한, CUT&RUN은 활용도가 높습니다. CUT&Tag의 경우, 타켓은 히스톤 PTM만 가능하지만, CUT&RUN은 다양한 타켓에 사용 가능합니다.
7. ChIP은 불용성 크로마틴 단백질은 분석할 수 없는데, CUT&RUN은 가능한가요?
네, 가능합니다. 사용한 논문은 다음과 같습니다.
Skene PJ, Henikoff S. An efficient targeted nuclease strategy for high-resolution mapping of DNA binding sites. Elife, 2017
8. CUT&RUN을 사용한 논문이 있나요?
네, CUT&RUN의 우수한 크로마틴 프로파일링 능력 덕분에 점점 많은 논문에서 CUT&RUN을 사용하고 있습니다. 5개의 우수한 논문을 알려드립니다.
- Yuan, Gang et al. Elevated NSD3 histone methylation activity drives squamous cell lung caner. Nature. 2021
- Yusufova, Nevin et al. Histone H1 loss drives lymphoma by disrupting 3D chromatin architecture. Nature. 2020
- Collins, Patrick et al. DNA double-strand breaks induce H2AX phosphorylation domains in a acontact-dependent manner. Nature Communications. 2020
- Sarthy et al. Histone deposition pathways determine the chromatin landscapes of H3.1 and H3.3 K27M oncohistones.eLife.2020
- Chen, Zhiyuan et al. Distinct dynamics and functions of H2AK119ub1 and H3K27me3 in mouse preimplantation embryos. Nature Genetics. 2021
또한, 아래의 논문들을 참고하면, CUT&RUN의 가장 기본적인 프로토콜 및 다양한 실험 목적에 적합한 프로토콜을 확인할 수 있습니다.
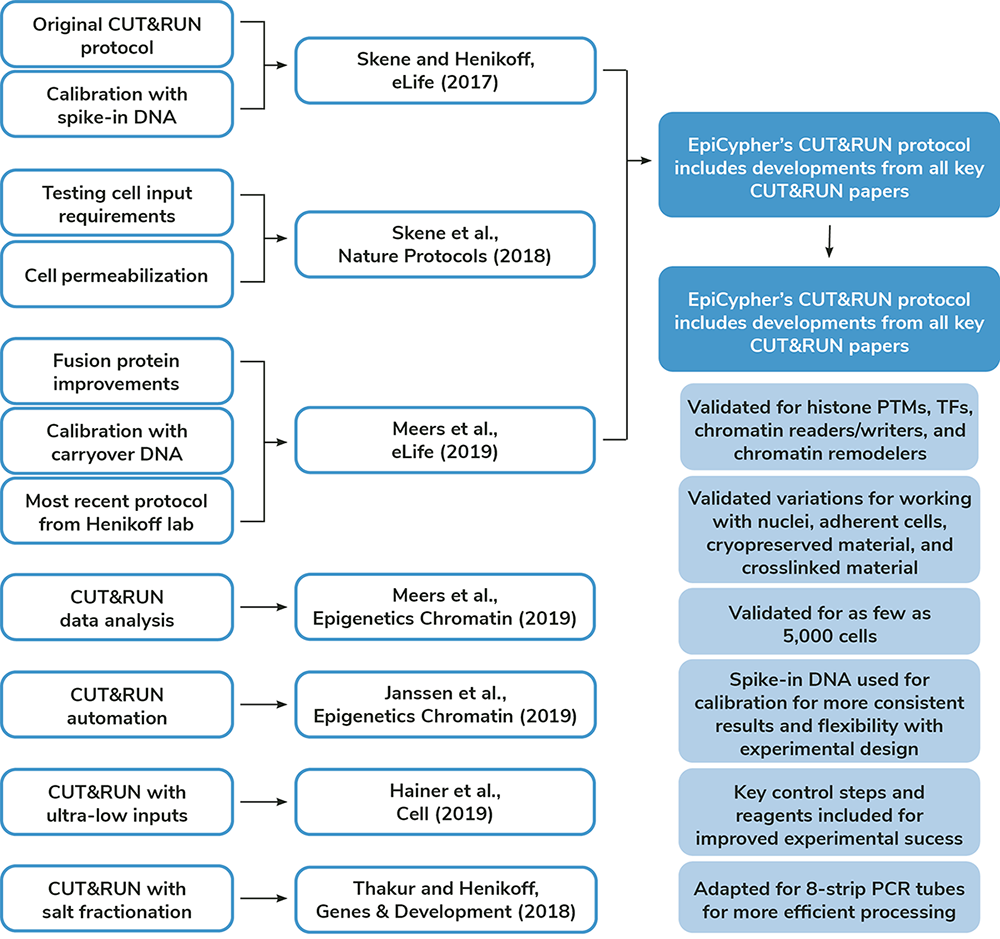
* 제품에 대한 상세 내용은 아래 추천 제품을 클릭해 주세요.