[안내] 이 서비스는 현재 이벤트 중입니다.
버전2 출시기념 20% 할인이벤트
[블로그] 더욱 향상된 새버전 CUT&Tag 출시! 무엇이 달라졌을까? 바로가기
CUTANA™ CUT&Tag Kit는?
Cleavage Under Targets & Tagmentation (CUT&Tag) 은 CUT&RUN과 ChIC 기술을 기반으로 하는 epigenomic mapping 기술입니다. CUTANA™ CUT&Tag에서는 핵을 고체 지지대에 고정하고 항체를 in situ로 chromatin target에 결합합니다. pAG-Tn5(단백질 A, 단백질 G와 prokaryotic transposase 5의 fusion)를 이용하여 선택적으로 절단하고 항체가 결합된 크로마틴을 시퀀싱 어댑터로 tagment합니다. Tagment된 조각은 EpiCypher의 "Direct-to-PCR"을 통해 단일 튜브에서 바로 증폭됩니다.
기존의 ChIP-seq에 비해, CUT&Tag은 민감도가 향상된 고품질의 시퀀싱 데이터를 제공합니다. Chromatin-fragmentation이나 IP 없이, 온전한 핵에서 항체가 라벨링된 크로마틴을 선별적으로 타겟팅하여 ChIP-seq에 비해 백그라운드가 확연히 줄어듭니다.
CUTANA™ CUT&Tag Kit는 민감도를 떨어뜨리지 않고 처리량 및 scale을 늘이고자 하는 genomic mapping 전문가에게 적합합니다. 하나의 튜브에서 진행되는 single-tube workflow는 multi-channel pipette에 적합하게 설계되었으며, 단 이틀만에 전 과정을 완료할 수 있습니다.
* chromatin-associated proteins (e.g. transcription factors)은 CUT&RUN kit 사용을 권장합니다.
특징
- 48번의 CUT&Tag reaction을 진행할 수 있는 구성품(pAG-Tn5, Con A bead, 버퍼, 튜브, 양성 대조군, 음성 대조군, spike-in control, indexing primer, PCR master mix, DNA 정제 bead)이 포함되어 있습니다.
- 세포에서 시퀀싱까지 단 이틀만에 진행할 수 있습니다.
- 독점적인 Direct-to-PCR를 이용하여, 하나의 튜브에서 세포부터 PCR 증폭 라이브러리를 만드는 전체 과정을 진행할 수 있습니다.
- 10,000 세포까지 신뢰할 수 있는 데이터를 얻을 정도로 민감도가 높기 때문에, rare cell type 및 low-input application에도 적합합니다.
- 5-8백만 시퀀싱만 읽어도 되므로, 분석 비용이 절감됩니다.
- 주요 양성 대조군, 음성 대조군, spike-in control이 포함되어 있어, 실험 과정을 최적화하고 모니터링할 수 있습니다.
- Index primer, PCR master mix, DNA 정제 비드가 포함되어 있어 라이브러리 제작 및 multiplex 시퀀싱을 빠르게 진행할 수 있습니다.
- 백그라운드에 비해 높은 시그널을 보입니다.
- Multichannel-pipette과 호환되어, 샘플 핸들링에 따른 변이를 줄입니다.
Kit 구성품
| *Kit
Version 3 기준 |
14-1102 |
14-1103 |
| Component |
Cat. No. |
Cat. No. |
| CUTANA™
pAG-Tn5 for CUT&Tag |
15-1017 |
15-1017 |
| H3K27me3
Antibody, SNAP-Certified™ for CUT&RUN and CUT&Tag |
13-0055t |
13-0055t |
| H3K4me3
Positive Control Antibody |
13-0060t |
13-0060t |
| CUTANA™
Rabbit IgG CUT&RUN Negative Control Antibody |
13-0042t |
13-0042t |
| Anti-Rabbit
Secondary Antibody for CUTANA™ CUT&Tag Workflows |
13-0047t |
13-0047t |
| SNAP-CUTANA™
K-MetStat Panel |
19-1002t |
19-1002t |
| CUTANA™
Concanavalin A Conjugated Paramagnetic Beads |
21-1401 |
21-1401 |
| CUTANA™
Non-Hot Start 2X PCR Master Mix for CUT&Tag |
15-1018 |
15-1018 |
| CUTANA™
CUT&RUN 8-strip 0.2 mL Tubes |
10-0009t |
10-0009t |
| 4.5 M NaCl |
21-1013 |
21-1013 |
| 0.5 M EDTA |
21-1014 |
21-1014 |
| 1 M MgCl2 |
21-1015 |
21-1015 |
| SDS Release
Buffer |
21-1017 |
21-1017 |
| SDS Quench
Buffer |
21-1018 |
21-1018 |
| 0.1X TE
Buffer |
21-1019 |
21-1019 |
| Pre-Wash
Buffer |
21-1020 |
21-1020 |
| Pre-Nuclear
Extraction Buffer |
21-1021 |
21-1021 |
| Bead
Activation Buffer |
21-1022 |
21-1022 |
| 5% Digitonin |
21-1023 |
21-1023 |
| 1 M
Spermidine |
21-1024 |
21-1024 |
| SPRIselect
reagent manufactured by Beckman Coulter, Inc |
21-1404 |
21-1404 |
| Multiplexing
Primers |
Primer Set 1 |
Primer Set 2 |
프로토콜
1. Magnetic ConA beads에 Nuclei 를 고정시킵니다.
2. 타겟 PTM에 대한 1차 & 2차 항체를 반응시킵니다.
3. pA-Tn5 (transpose)와 Mg2+를 처리하여 항체가 붙은 타겟 크로마틴을 절단하고 시퀀싱 어댑터를 tagging 합니다.
4. PCR로 DNA를 증폭합니다.
5. NGS로 시퀀싱을 진행합니다.
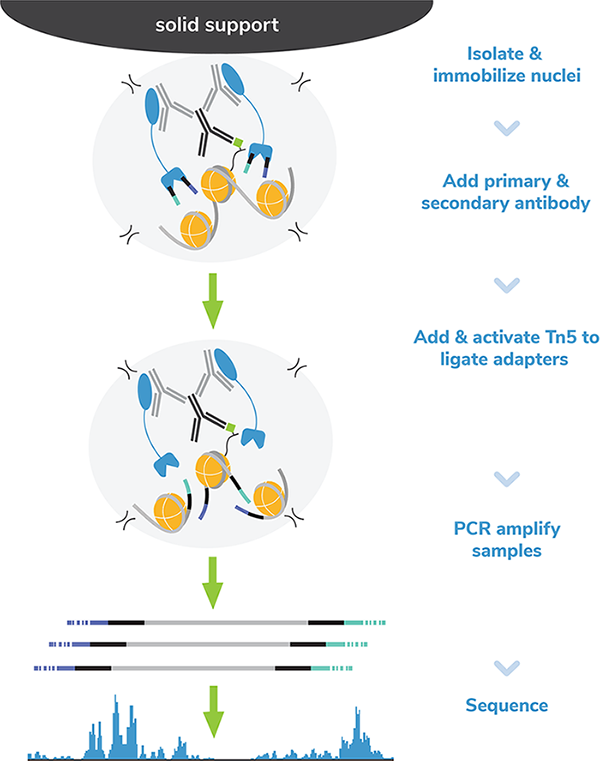
실험과정
Day 1-2) CUTANA™ CUT&Tag Kit을 이용하여 NGS를 위한 library를 준비합니다.
Day 2-3) NGS 시퀀싱을 진행합니다.
Day 3) 데이터를 분석합니다.

참고 데이터
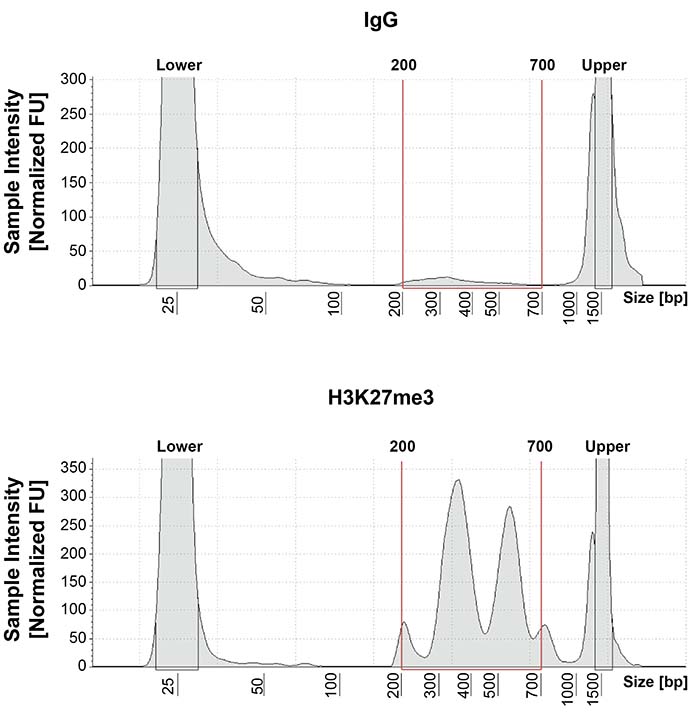
1. CUT&Tag DNA fragment size distribution analysis
CUT&Tag을 수행한 결과 데이터입니다. Library DNA는 Agilent TapeStation®으로 분석되었으며, 이를 통해 mononucleasome은 300-400bp 사이의 peak에서, dinucleasome은 500-700bp 사이의 peak에서 관찰되었습니다.
2. SNAP-CUTANA™ K-MetStat Spike-in controls
16가지 서로 다른 K-메틸 PTM 상태 (mono-, di-, tri-methylation at H3K4, H3K9, H3K27, H3K36, H4K20, unmodified control)의 dNucs을 CUT&Tag 샘플에 스파이크하였고, spike-in control로 이용되는 SNAP-CUTANA K-MetStat Panel (#19-1002)로 최적의 실험조건을 확인하였습니다.
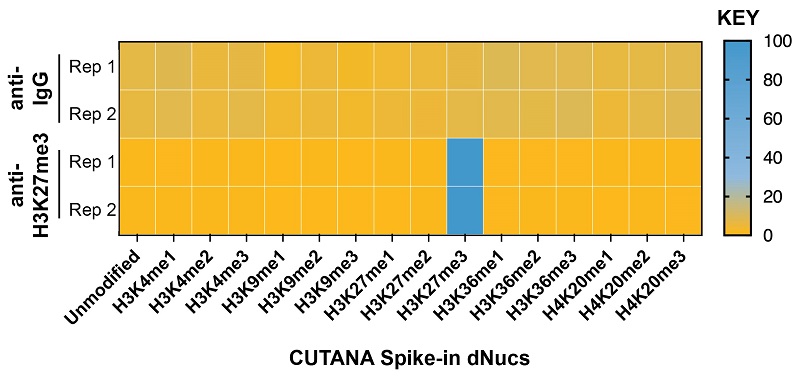
DNA-barcoded designer nucleosomes (dNucs) representing 16 different K-methyl PTM states: mono-, di-, and tri-methylation at H3K4, H3K9, H3K27, H3K36, and H4K20, as well as unmodified control, were spiked into CUT&Tag samples prior to the addition of the control antibodies provided with the kit (IgG, H3K27me3). After sequencing, instances of each spike-in barcode recovered in the CUT&Tag reactions were counted and normalized from raw fastq files using the shell script and analysis excel sheet available on the spike-in product page (EpiCypher 19-1002). Barcodes for IgG (top; normalized to the sum of total reads) and H3K27me3 (bottom; normalized to on-target) antibodies provided with this kit are shown. The spike-ins confirmed optimal experimental conditions (H3K27me3 antibody specifically recovered the target dNuc, while IgG showed no preferential enrichment).
3. CUT&Tag genome-wide heatmaps
CUT&Tag 수행 결과를 히트맵으로 표시한 결과입니다.
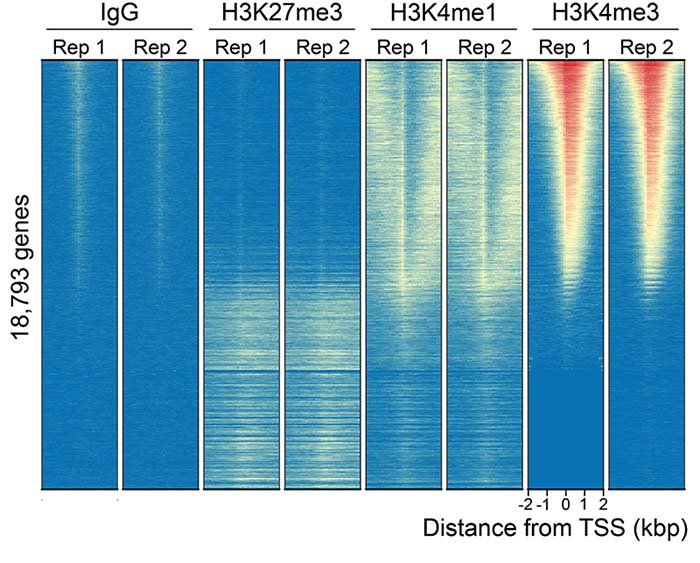
CUT&Tag was performed as described above. Heatmaps show two replicates (“Rep”) of IgG, H3K27me3, H3K4me1, and H3K4me3 antibodies in aligned rows ranked by intensity (top to bottom) relative to the H3K4me3 Rep 1 reaction. High, medium, and low intensity are shown in red, yellow, and blue, respectively. Antibodies to histone PTMs showed expected enrichment patterns and high reproducibility. H3K4me3, a marker of active transcription localized to TSSs, shows oppositional enrichment to H3K27me3 (a marker of repressive chromatin), while H3K4me1 signal flanks TSSs. IgG shows low background enrichment.
함께 이용하는 제품
- ConA bead washing : Magnetic Separation Rack (보러가기)
- CUT&RUN Antibody (보러가기)
- SNAP-ChIP antibody & spike-in control (보러가기)
ChIP-seq vs. CUT&RUN vs. CUT&Tag 비교
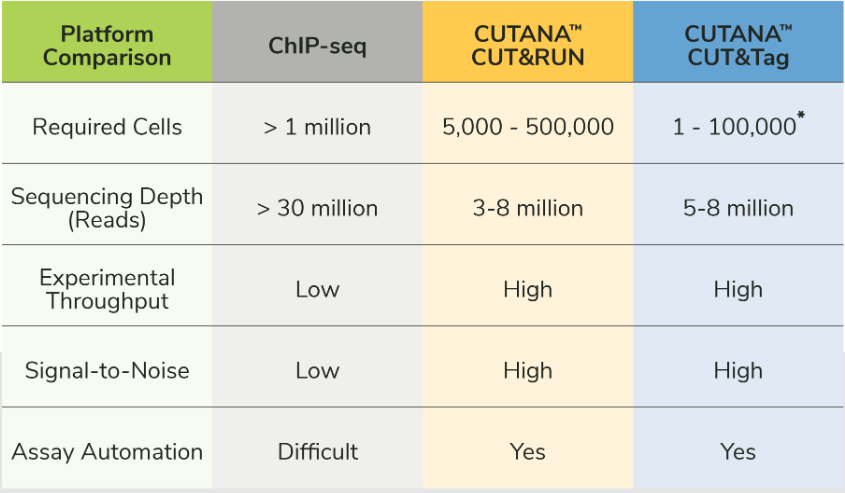
*CUT&Tag has been applied for single-cell chromatin analysis in the literature. However, the recommended input for CUTANA™ CUT&Tag assays is 10,000 to 100,000 cells.
주요 사용 논문
Kaya-Okur et al. CUT&Tag for efficient epigenomic profiling of small samples and single cells. Nat. Comm. 10, 1930 (2019). (PMID: 31036827)
This paper introduces CUT&Tag methodology, as developed by the Henikoff laboratory at the Fred Hutchinson Cancer Research Center. It describes how the Tn5 fusion proteins works, demonstrates the application of CUT&Tag for histone PTM and chromatin factor profiling, and reveals major advantages of this new technology. Specifically, they use CUT&Tag to generate high-quality histone PTM profiles from single cells, a major advancement for the epigenetics field.
Kaya-Okur et al. Efficient low-cost chromatin profiling with CUT&Tag. Nat. Protoc. 15, 3264-3283 (2020). (PMID: 32913232)
Here, the inventors of CUT&Tag provide a highly detailed description of their optimized CUT&Tag protocol, citing EpiCypher’s pAG-Tn5 as the suggested commercial vendor of the enzyme. The protocol highlights critical steps, includes a troubleshooting FAQ table, and shows example results.
Henikoff et al. Efficient transcription-coupled chromatin accessibility mapping in situ. by nucleosome-tethered tagmentation. Elife 16;9:e63274.(2020). (PMID: 33191916)
The inventors of CUT&RUN / CUT&Tag technology leveraged EpiCypher’s pAG-Tn5 and CUT&RUN compatible histone PTM antibodies to develop a modified version of CUT&Tag for chromatin accessibility mapping. Importantly, this new method generates robust, high-quality profiles using only 3-5M reads/samples (vs. 20-30M reads for standard ATAC-seq).
Janssens et al. Automated CUT&Tag profiling of chromatin heterogeneity in mixed-lineage leukemia. Nat. Genet. 53, 1586-1596 (2021). (PMID: 34663924)
Assay automation is a major feature of CUT&Tag, as this will enable higher throughput for clinical research projects. In this study, Janssens et al. develop an automated CUT&Tag platform and use it to map oncofusion proteins, transcription factors, and histone PTMs in samples from leukemia patients. They also modified their approach for single cell analysis to assess inter- and intra-tumor heterogeneity.
Janssens et al. CUT&Tag2for1: a modified method for simultaneous profiling of the accessible and silenced regulome in single cells Genome Biol. 23, 81.(2022). (PMID: 35300717)
CUT&Tag2For1 is an innovative application of CUT&Tag profiling technology that allows simultaneous, high-resolution mapping of transcription initiation (RNA Pol II phospho S5) and repressive domains (H3K27me3) in single cells or bulk populations. Mapping multiple chromatin features in a single reaction is important when cell numbers are limiting, including biopsy studies and clinical research.
논문 더 보기